430685
430685

实验目的
为了获取能与岩沙海葵毒素(Palytoxin,PLTX)特异性结合的天然适配体,目标细胞内提取得到的基因组通过超声打断的方式变成 100-300 bp 的 DNA 片段,再通过冷淬的方式获得折叠后的 ssDNA。将 ssDNA 进行甲基化修饰后进行测序,同时对甲基化修饰后的片段进行亲和力测定,最终获得特异性强、亲和力高的天然适配体。
实验步骤
HaCaT 和 CHO-K1 细胞基因组 DNA 的提取:
①常规培养细胞,待其融合度达到 70% - 80%左右时,吸除原培养基,向每 10 cm2的贴壁细胞中加入 1 mL PBS,用移液枪吹落细胞,转移至 1.5 mL EP 管中,5000 rpm 离心 5 min,弃上清,加入 200 μL 的 PBS 重悬细胞;
②按照 TaKaRa MiniBEST Universal Genomic DNA Extraction Kit 说明,加入 180 μL Buffer GB、20 μL Proteinase K 和 10 μL RNase A,充分吹打混匀,于 56℃水浴 10 min;
③向裂解液中加入 200 μL 100%乙醇,充分吹打混匀后,将其转移至已经安置于 Collection Tube 上的 Spin Column 内,12000 rpm 离心 2 min,弃滤液;
④将 500 μL Buffer WA 加入至 Spin Column 中,12000 rpm 离心 1 min;
⑤将 700 μL Buffer WB 加入至 Spin Column 中,12000 rpm 离心 1 min,并重复 1 次;
⑥将 Spin Column 安置于 Collection Tube 上,12000 rpm 离心 2 min;
⑦将 Spin Column 安置于新的 1.5 mL EP 管上,在 Spin Column 膜的中央处加入 50-200 μL 已加热至 65℃的 Elution Buffer,室温静置 5 min;
⑧12000 rpm 离心 2 min,洗脱 DNA;
⑨使用 NanoDropTM One 进行基因组 DNA 定量。
片段化基因组 :将提取的基因组 DNA 放置于SCIENTZ08-IIIC非接触式超声波细胞粉碎机中,设置功率80%,超声开时间为 1 s,超声关时间为 3 s,工作总时间为 150 min,温度为 15℃,片段化基因组 DNA。
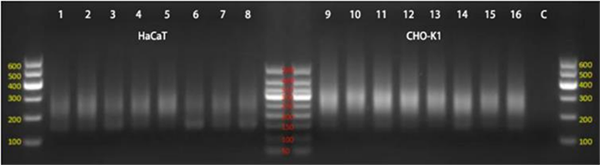
核酸凝胶电泳 :利用核酸凝胶电泳检测 DNA 片段的大小,核酸凝胶的配方为:ddH2O 6.69 mL、40%聚丙烯酰胺 1.31 mL、5×TBE 2.4 mL、10%过硫酸铵(Ammonium persulfate,AP)72μL、四甲基乙二胺(Tetramethylethylenediamine,TEMED)18 μL。在样品中加入 5×上样缓冲液,充分混合均匀,将配置好的凝胶放于电泳槽中,加入 1×TBE 电泳缓冲液,垂直拔出梳子,立刻用缓冲液冲洗加样孔,将样品缓慢均匀加入孔中,300 V 电泳 20 min,取出凝胶放置于水平摇床,用 Gel-red 染色液避光孵育 20 min,在凝胶成像仪中成像,检测 DNA 片段大小范围。
实验结果
基因组来源的 ssDNA 文库的评价
超声破碎基因组后,利用核酸凝胶电泳检测基因组片段大小。如图所示,DNA 片段长度富集于 200 - 300 bp 之间,当其猝冷变性折叠成单链后,ssDNA 长度则集中于 200-300 nt 之间,从 HaCaT 和 CHO-K1 细胞来源的基因组文库中各取 18 nmol 的 ssDNA 用于后续实验。
结论
结论:提取得到的细胞基因组经过超声打断后得到 200-300 bp 大小的片段,可直接用于高通量测序,也可重硫酸盐转化后用于甲基化测序。
24小时服务热线 18106650612